Índice
- 1 Vigilancia epidemiológica del sarampión: RT-PCR en tiempo real
- 2 Tipo de muestras para el examen
- 3
- 4 Diagnóstico mediante RT-PCR en tiempo real
- 5 Control de calidad para la RT-PCR en tiempo real para el diagnóstico y vigilancia epidemiológica del sarampión
- 6 Rango reportable del ensayo
- 7 Tu opinión es importante
- 8 Fuentes
Vigilancia epidemiológica del sarampión: RT-PCR en tiempo real
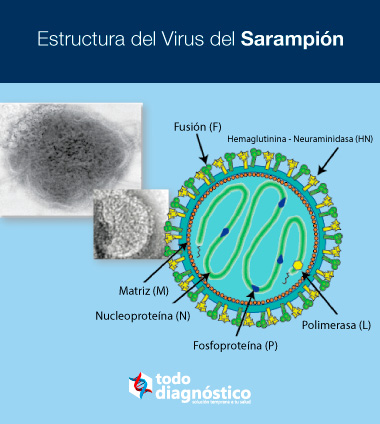
El virus de sarampión es un virus de ARN monocatenario miembro de la familia de los Paramixovirus, cuyo único huésped es el ser humano. Su genoma consta de 15,894 nucleótidos que se codifican para seis proteínas estructurales: nucleoproteína (N), fosfoproteína (P), matriz (M), fusión (F), hemaglutinina (H), polimerasa (L) y dos proteínas no estructurales: C y V. Los síntomas principales del sarampión son fiebre, exantema, tos, coriza y/o conjuntivitis. Debido al disparo en el número de casos de sarampión a nivel global, es importante hablar de la vigilancia epidemiológica del sarampión.
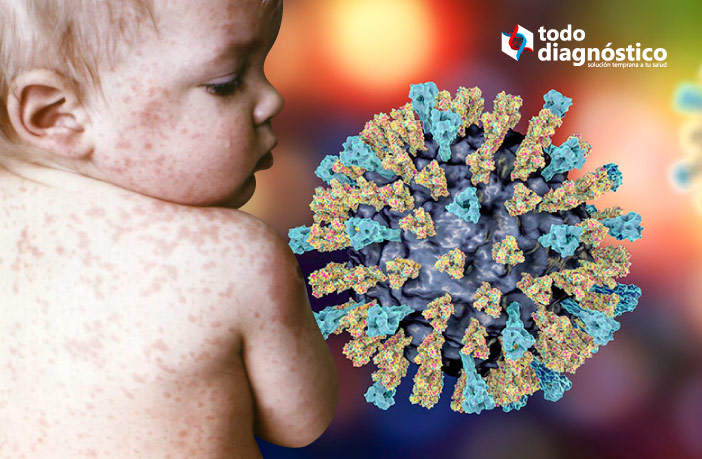
 En la década de 1990 la Organización Mundial de la Salud (OMS) señaló que el mundo estaba a un paso de la eliminación del sarampión, una enfermedad altamente infecciosa. Hoy esta perspectiva se ve seriamente amenazada por el resurgimiento de este padecimiento en varios países como Francia, España y Estados Unidos.
En la década de 1990 la Organización Mundial de la Salud (OMS) señaló que el mundo estaba a un paso de la eliminación del sarampión, una enfermedad altamente infecciosa. Hoy esta perspectiva se ve seriamente amenazada por el resurgimiento de este padecimiento en varios países como Francia, España y Estados Unidos.
“A nivel mundial, el sarampión sigue siendo una de las principales causas de muerte en niños pequeños a pesar de que existe una vacuna segura y eficaz. Se calcula que en 2016 murieron casi 90 mil personas por esta causa, la mayoría de ellas menores de 5 años”.
Para inicios de abril de 2020 en México suman 101 casos de sarampión. De los cuales, más del 90% se ubican en la Ciudad de México. No se había presentado brotes de sarampión significativos desde el año 2007. Existe el riesgo epidemiológico latente de la reintroducción del virus en todo el país debido al aumento que se ha ido observado. Por lo tanto, se debe mantener la vigilancia epidemiológica del sarampión en México. Esto mediante la identificación de casos probables que cumplen con la sintomatología descrita y confirmarlos mediante pruebas de laboratorio especializadas. La herramienta actual disponible para el diagnóstico del sarampión es la PCR. Esta prueba de diagnóstico detecta material genético del virus y técnicas serológicas que identifican anticuerpos anti-sarampión.
Tipo de muestras para el examen
El examen se puede realizar a partir de muestras de exudado faríngeo, orina y líquido cefalorraquídeo. Las muestras de exudado faríngeo deben estar contenidas en medio de transporte viral. Mientras que las muestras de orina y líquido cefalorraquídeo deben estar contenidas en un frasco estéril con tapón de rosca.
Las muestras deben ser tomadas durante los primeros cinco días inmediatos a la aparición del exantema y ser enviadas a refrigeración (4-8 °C) en un lapso no mayor de 24 horas después de haber sido tomadas.
Diagnóstico mediante RT-PCR en tiempo real
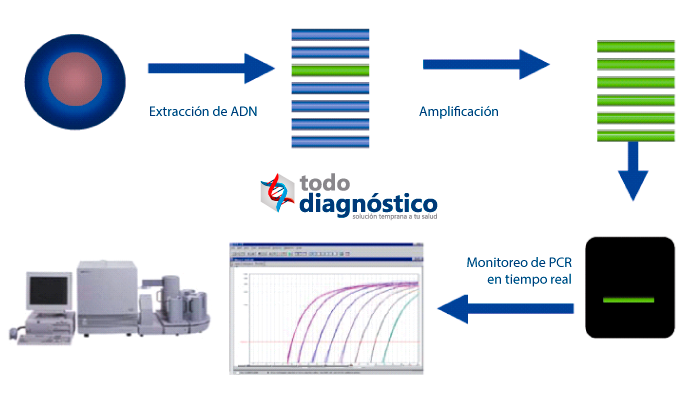
La reacción en cadena de la polimerasa (PCR) tiene la facultad de producir múltiples copias de un fragmento de ADN específico. En conjunto con la reacción de retrotranscripción (RT), es posible obtener moléculas de ADN a partir de ARN. El ARN extraído de muestras clínicas es convertido en ADN complementario (ADNc) y posteriormente amplificado para obtener un gran número de copias. A este método se le conoce como RT-PCR punto final, debido a que el producto final del último ciclo de amplificación es visualizado en gel de agarosa mediante electroforesis.
En cambio, la RT-PCR en tiempo real, es un método que tiene la capacidad de monitorear el progreso de la amplificación ciclo por ciclo. La detección de la amplificación de un gen blanco es monitoreada mediante la captación de señales de fluorescencia emitida durante los ciclos de la PCR. Para la detección del virus de sarampión mediante esta metodología se utilizan sondas TaqMan.
“En la RT-PCR en tiempo real el gen blanco codifica una de las 6 proteínas estructurales del virus de sarampión para la nucleoproteína (N) ”
Una sonda TaqMan es una secuencia nucleotídica de 20 a 30 bases de longitud aproximadamente, que tiene un fluorocromo unido covalentemente en el extremo 5’, llamado fluorocromo “reportero”; así como un apagador o “quencher” en el extremo 3’. Cuando la sonda hibrida con su secuencia blanco entre el iniciador sentido y anti sentido, el fluorocromo es liberado por medio de la actividad 5′ exonucleasa de la ADN polimerasa. Este sistema permite monitorear el cambio en el patrón de fluorescencia y deducir el nivel de amplificación del gen blanco ciclo por ciclo, en tiempo real.
Cuando la sonda está intacta, se presenta una transferencia energética de fluorescencia por resonancia FRET (del inglés “Frequence Resonance Energy Transference”) desde el fluorocromo reportero, al “quencher”.
Control de calidad para la RT-PCR en tiempo real para el diagnóstico y vigilancia epidemiológica del sarampión
Para la vigilancia epidemiológica del sarampión y para verificar la fidelidad de los resultados en cada ensayo de RT-PCR en tiempo real se incluye:
- Un control negativo o NTC (del inglés no template control), comúnmente es agua grado biología molecular.
- Un control negativo de extracción (CNE), para verificar la integridad del reactivo de extracción de ARN. Se agrega agua o cualquier otra muestra que no sea un caso probable de sarampión.
- Un control positivo, que es material genético del virus de sarampión.
- Un control de muestra humana (HSC), que proporciona una señal positiva en el ensayo y demuestra una recuperación exitosa de ARN, así como la integridad del reactivo de extracción de ARN. En este ensayo se busca el ARN de la RNasa P (RP) humano, que está presente en la mayoría de las muestras clínicas. Es detectable mediante RT-PCR en tiempo real usando los iniciadores y sondas proporcionados.
Si existe alguna falla en el control de calidad del ensayo, este debe repetirse bajo las mismas condiciones.
Rango reportable del ensayo
Los valores de amplificación se expresan en valores de Ct (del inglés Cycle Threshold), donde un Ct es el ciclo a partir del cual los valores de fluorescencia relativa (UFR) rebasan el punto de corte o umbral y es inversamente proporcional a la cantidad de ARN o ADN. La amplificación se observa como una curva sigmoide y los posibles resultados obtenidos son:
- Positivo: significa que existe material genético del virus de sarampión (se observa una curva sigmoide).
- Negativo: quiere decir que no existe material genético del virus, por lo tanto no se observa curva o se observa una curva sigmoide de amplificación tardía con valor de Ct ≥: 38.
- Muestra inadecuada: puede ocurrir por diferentes razones. Por ejemplo, no hubo amplificación del control positivo, se observó curva en el control negativo (NTC), se observó amplificación en el control negativo de extracción, o no hubo amplificación del control de muestra humana (HSC).
Los resultados y la información demográfica de los pacientes deben reportarse para una óptima vigilancia epidemiológica del sarampión.
 Tu opinión es importante
Tu opinión es importante
Nuestro objetivo en Todo diagnóstico es informarte y ayudarte a tomar las mejores decisiones acerca de lo más valioso que tienes, tu salud y la de tus seres queridos. Hacemos un gran esfuerzo por presentar información de manera sencilla y fácil de entender. Finalmente, ayúdanos a saber si estamos haciendo bien nuestro trabajo. Califica nuestro artículo y/o deja un breve comentario.
Fuentes
OMS. (2006). Manual para el diagnóstico de laboratorio de la infección por los virus del sarampión y de la rubéola. 2006, de OMS Sitio web: https://www.who.int/ihr/elibrary/manual_diagn_lab_mea_rub_sp.pdf
Luis Delpiano, Leonor Astroza y Jorge Toro. (2015). Sarampión: la enfermedad, epidemiología, historia y los programas de vacunación en Chile. 2015, de Revista chilena de infectología Sitio web: https://scielo.conicyt.cl/pdf/rci/v32n4/art08.pdf
Juan Emilio Echevarría, Aurora Fernández García y Fernando de Ory. (2015). Vigilancia microbiológica del sarampión y la rubéola en España. Red de laboratorios. 2015, de Revista española de salud pública Sitio web: http://scielo.isciii.es/scielo.php?script=sci_arttext&pid=S1135-57272015000400006
CDC. (2018). Sarampión. 2018, de CDC Sitio web: https://www.cdc.gov/measles/about/signs-symptoms-sp.html
Redacción Animal Político. (2020). Suman 101 casos de sarampión en Valle de México; aparece un contagio en Campeche. 1 de abril de 2020, de Animal Político Sitio web: https://www.animalpolitico.com/2020/04/sarampion-aumento-casos-valle-mexico-campeche/
¡Califica nuestro artículo!
Ayúdanos a mejorar. Califica nuestro artículo para que sepamos si te fue útil y es de tu agrado, o bien, podamos mejorarlo para lectores futuros. ¡Gracias!





2 comentarios
Es un buen articulo
Gracias por los comentarios. Saludos!